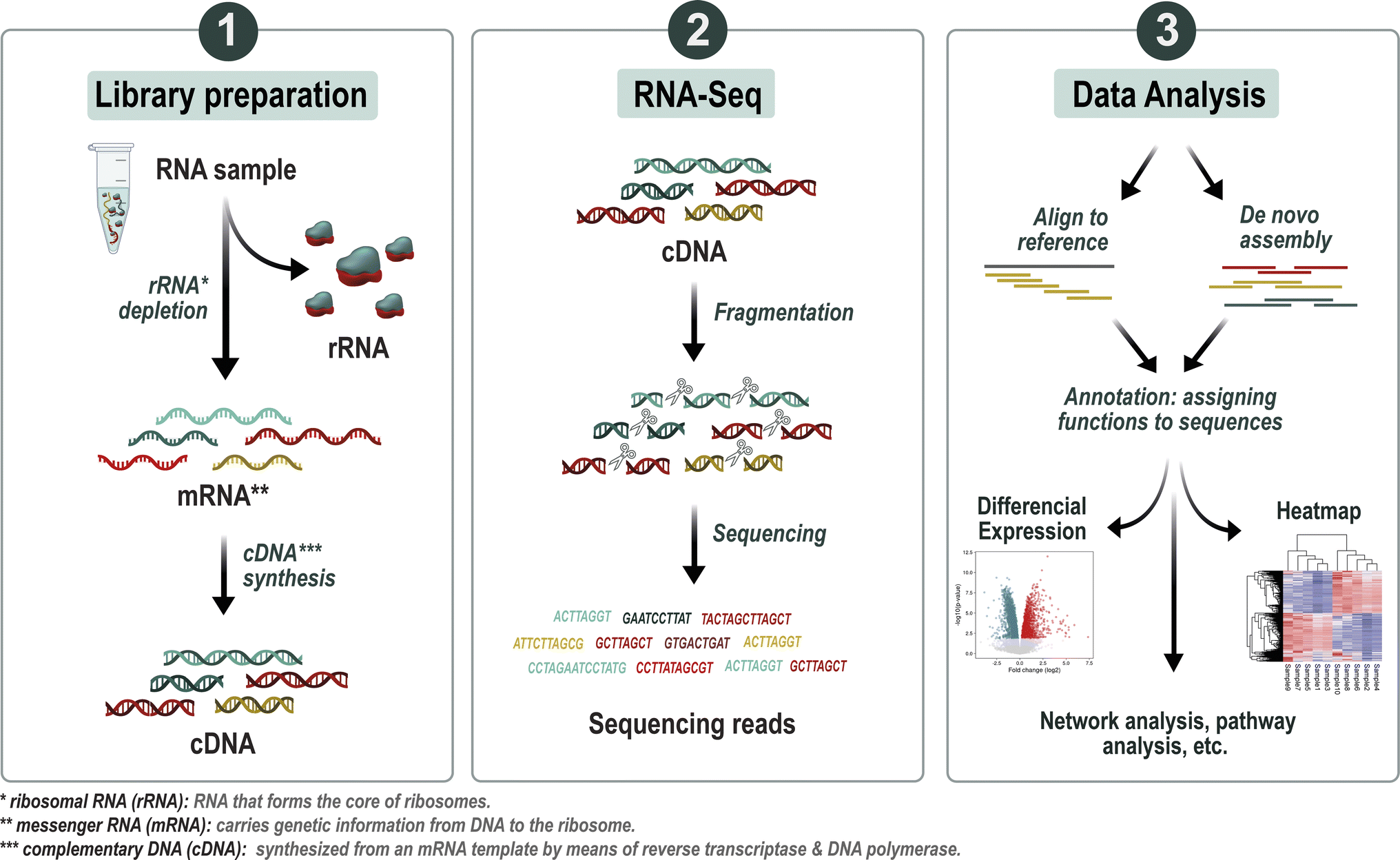
Métatranscriptomique
La méta-transcriptomique permet d’analyser les ARN messagers exprimés au sein de communautés microbiennes complexes, révélant ainsi l’activité fonctionnelle plutôt que la simple présence génétique des microorganismes (Zang et al., 2021).
Étapes expérimentales
a) Collecte & extraction, échantillonnage respectant la stabilisation rapide des ARN pour éviter la dégradation. Extraction d’ARN total depuis l’échantillon (sol, eau, flore intestinale…), qui contient majoritairement du rRNA.
b) Déplétion des ARNr (rRNA/tRNA) via kits commerciaux ou capture par billes, éliminant jusqu’à 70–85 % des ARNr.
c) Fragmentation & création de librairie
- Conversion de l’ARNm en cDNA par transcription inverse avec des protocoles strand-specific, souvent pour plateformes Illumina offrant des lectures courtes à haut débit (≥100 M lectures par échantillon), préparation des librairies : ligation d’adaptateurs, amplification PCR.
- Autre option : séquençage direct de l’ARN par nanopores (Oxford Nanopore), long reads, sans PCR, sans biais d’assemblage, mais avec un taux d’erreurs plus élevé.
d) Séquençage à haut débit, Illumina, Element BioSciences ou MGI pour lectures courtes ; Oxford Nanopore pour lectures longues, offrant un accès à des transcrits complets ou isoformes.
e) Analyse bioinformatique
- Contrôle qualité & filtrage, utilisation d’outils comme FastQC, Trimmomatic, SortMeRNA pour écarter les lectures de faible qualité ou dérivées de rRNA/tRNA.
- Assemblage / Mapping, Assemblage des lectures (de novo ou par mappage sur bases de référence), affectation taxonomique via outils comme Kraken2, BLASTN, mapping sur SILVA, Greengenes ou bases génomiques.
- Annotation fonctionnelle, Identification des ORFs, annotation via bases KEGG, MetaHIT, M5nr, COG, GO, VFDB ou CARD selon requête (antibiorésistance, virulence, etc.) → assignation des fonctions biologiques.
Nouvelle technique : Utilisation du séquençage direct ARN par nanopore pour accéder à des transcripts longs, isoformes et ARN structuraux sans cDNA, en cours d’optimisation.
Zhang Y, et al. Metatranscriptomics for the Human Microbiome and Microbial Community Functional Profiling, Annual Review of Biomedical Data Science Vol. 4:279-311 (juillet 2021) https://doi.org/10.1146/annurev-biodatasci-031121-103035
Esser M, Brinkmann M and Hecker M, Solving freshwater conservation challenges through next-generation sequencing approaches, DOI: 10.1039/D4VA00112E (Perspective) Environ. Sci.: Adv., 2024, 3, 1181-1196, https://pubs.rsc.org/en/content/articlehtml/2024/va/d4va00112e