Atlas single cell du poumon entier
Le premier atlas intégré des cellules pulmonaires humaines révèle des informations sur les maladies pulmonaires
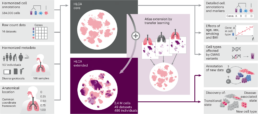
La carte cellulaire la plus grande et la plus complète du poumon humain a récemment été publiée dans Nature Medicine (Lien). Révélant la grande diversité des types cellulaires pulmonaires et les principales différences entre être en bonne santé et être malade, l’Atlas des cellules pulmonaires humaines sera une ressource précieuse pour les chercheurs.
En combinant les données de 2,4 millions de cellules décrites dans ce dataset provenant de 486 individus et 49 ensembles de données, les chercheurs ont créé le premier atlas monocellulaire intégré du poumon, révélant des types de cellules rares et mettant en évidence les différences cellulaires entre les personnes en bonne santé. De plus, l’étude a trouvé des états cellulaires communs entre la fibrose pulmonaire, le cancer et le COVID-19, offrant de nouvelles façons de comprendre les maladies pulmonaires, ce qui pourrait aider à identifier de nouvelles cibles thérapeutiques.
Le « Human Cell Atlas Lung Biological Network » est un groupe de scientifiques qui collaborent pour cartographier les types et états cellulaires présents dans les voies respiratoires humaines. Ce groupe est coordonné entre autres par Pascal Barbry, responsable d’UCAGenomiX, Nice et France Génomique a contribué au projet.