Plateforme de séquençage de Paris-Saclay
La Plateforme transcriptOmique de Paris Saclay, POPS, localisée à Gif sur Yvette (91), a été créée en 2003 et a pour mission principale de proposer un panel de prestations techniques et bio-informatiques pour l’analyse du transcriptome des plantes. C’est le « service intégré à façon ».
POPS permet à une communauté scientifique publique ou privée de répondre à des questions biologiques grâce au séquençage à haut débit des ARNs couramment nommé le RNA-Seq. Nous réalisons des projets d’étude en collaboration avec des équipes de recherche nationales et internationales. Nous avons fait le choix de nous spécialiser exclusivement sur les végétaux afin d’avoir une expertise forte et de conseiller au mieux nos collaborateurs sur leurs projets. A ce jour, nous avons travaillé sur plus de 93 espèces différentes.
Notre champ d’application implique de rester à la pointe de la technologie dans le domaine du séquençage et de l’analyse des données afin de proposer des protocoles techniques et des analyses bioinformatiques les plus pertinents et intéressants tant sur le plan biologique que financier, notre seconde mission est le « développement ».
Enfin transmettre le savoir acquis grâce à la communauté est important. La plateforme dispense des formations, participe à des enseignements, coorganise les « journées omiques végétales » et accueille des stagiaires. C’est un aspect essentiel nommé « Formation & animation scientifique ».
Prestations
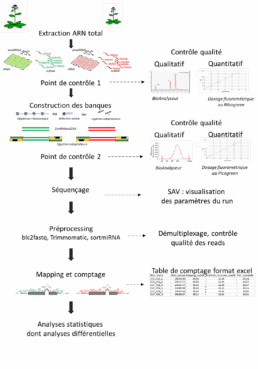
Riche de son expertise dans les végétaux, POPS propose différents protocoles et analyses pour réponse aux questions biologiques des utilisateurs. Les différentes étapes pour mener un projet sont les suivantes :
(*Ces étapes peuvent être prises en charge par les collaborateurs.)
- Discussion du design expérimental et choix de la technologie la plus adaptée,
- Broyage des échantillons*,
- Extractions des ARNs*,
- Contrôle qualitatif et la quantitatif des ARNs,
- Construction des banques,
- Contrôle qualitatif et quantitatif des banques,
- Séquençage,
- Analyse des données*,
- Interprétations des résultats*,
- Prises en charge de la soumission dans les bases de données*.
Expertises
N’hésitez pas à les contacter pour des protocoles personnalisables en fonction de besoins spécifiques.
- Séquençage orienté des ARN polyAdenylés ou non
- Séquençage des petits ARN : miRNA, siRNA …
- Séquençage de longs fragments (projets collaboratifs)
- Microtranscriptomique ou séquençage de très faible quantité d’ARN (à partir de 75pg d’ARN total).
- Métatranscriptomique ou séquençage du transcriptome de plusieurs espèces à partir d’un même échantillon.
- Empreintes
- Cellule unique (10X, Rhapsody, ScaleBiosciences)
- Criblage transcriptomique à grande échelle (BRB-Seq)
Bio-informatique et statistiques
Les services de POPS peuvent inclure les analyses bioinformatiques et statistiques des données générées. Ceci est possible grâce à l’implication de l’équipe « Réseaux génomiques » de l’IPS2 connue pour ses développements de méthodes et de pipelines pour les données omiques.
Une analyse bioinformatique et statistique faite par POPS assure que les données sont privées durant une période décidée avec le collaborateur puis rendues publiques grâce au dépôt des données dans la base CATdb (site) et la base internationale du NCBI (GEO/SRA).
Dans le cadre du « service intégré », POPS propose systématiquement un pré-processing pour s’assurer de la qualité des données et le stockage durant 6 mois après envoi aux collaborateurs (dans la limite de 1 To). Ce service peut être complété par d’autres services tels que :
- Une analyse bioinformatique par échantillon pour fournir une table de comptage
- Une analyse bioinformatique et statistique du RNA-seq classique (analyses différentielles, analyses de coexpression, tests d’enrichissements, étude des éléments cis-régulateurs de la transcription)
- Une analyse bioinformatique et statistique du single-cell (identification des clusters de cellules, des biomarqueurs et analyses differentielles)
- Une analyse bioinformatique et statistique pour le criblage transcriptomique.
- Une construction d’un transcriptome de référence (génome de référence non accessible) et analyse bioinformatique et statistique
- Assemblage et annotation de transcriptome de novo (dont Nanopore)
Equipements
- Contrôle qualité : Bioanalyzer (Agilent), Fragment Analyser (Agilent), CLARIOstar Plus (BMG Labtech), Qubit (Invitrogen), Pippin HT (Sage Science)
- Trieur de cellules : MACSQuant tyto (Miltenyo Biotec)
- Séparateur de cellules : Chromium (10X Genomics), HT Express Rhapsody (BD Sciences)
- Robot pipeteur : Biomek FX (Beckman)
- Séquenceurs : NextSeq500 (Illumina), NovaSeq (Prestataires extérieurs), MinION (Nanopore), P2 (Nanopore)
- Sonicateur : M220 Covaris (LGS Genomics)
- Informatique : Serveur (DELL), Station de calcul (DELL)
Principales réalisations
Mise à jour : Aout 2025

Certification / Assurance qualité
Certifiée ISO9001 depuis 2012
Labels
Plateforme IBiSA depuis 2008
Plateforme Stratégique Nationale par l’INRAE depuis 2008



Responsables de la Plateforme
Responsable scientifique
Etienne Delannoy
Responsable opérationnelle
Médine Benchouaïa
IPS2, Institute of Plant Sciences – Paris-Saclay
Bâtiment 630, avenue des Sciences
Plateau du Moulon
91190 – Gif-sur-Yvette
Contacts
Etienne Delannoy,
Médine Benchouaïa
pops.ips2@universite-paris-saclay.fr
POPS, Plateforme TranscriptOmique de l’iPS2
01 69 15 77 19

